研究内容(トピックス)
紫外線により傷ついたDNAの修復メカニズムを解明
- Cavadini, S., Fischer, E. S., Bunker, R. D., Potenza, A., Lingaraju, G. M., Goldie, K. N., Mohamed, W. I., Faty, M., Petzold, G., Beckwith, R. E., Tichkule, R. B., Hassiepen, U., Abdulrahman, W., Pantelic, R. S., Matsumoto, S., Sugasawa, K., Stahlberg, H., and Thomä, N. H. (2016) Cullin-RING ubiquitin E3 ligase regulation by the COP9 signalosome. Nature 531: 598-603.
DNAの損傷は、1日のうち1細胞あたり数万回以上も発生するとされ、修復が間に合わなくなるとDNAの複製・転写の妨害、細胞死や染色体の不安定化を引き起こし、さらには細胞ががん化する恐れもあります。したがって、DNA修復は、細胞が正常な機能を発揮し、生存するために必要不可欠な機能であるといえます。
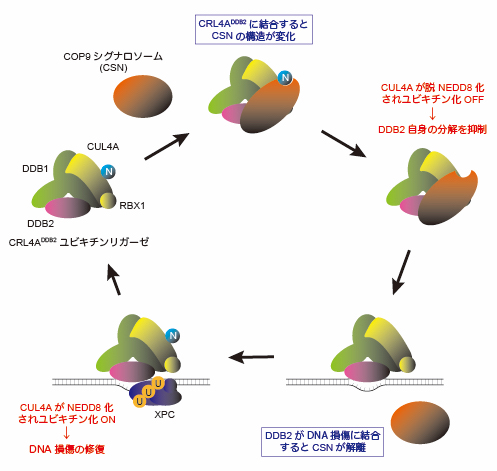
DNA損傷を引き起こす要因の一つとして紫外線が挙げられます。紫外線によりDNAが損傷すると、「DDB1-CUL4A-RBX1(CRL4A)」に「DDB2」が結合したタンパク質複合体(CRL4ADDB2ユビキチンリガーゼ)が損傷を見つけて結合し、周囲のタンパク質のユビキチン化※1を引き起こすことがDNA修復のスムーズな進行に重要です。この時、CRL4Aを構成するCUL4Aに「NEDD8」という小さなタンパク質が結合することが、ユビキチン化の活性化に必要であることがわかっていました。一方で、DNAが損傷していない場合には、「COP9シグナロソーム(CSN)」と呼ばれるタンパク質複合体のはたらきによってNEDD8が取り除かれ、CRL4Aが不活性化されています。しかし、DNA修復機構で重要なプロセスであるユビキチン化がどのように制御されているのか、詳細なメカニズムはこれまで不明でした。
私たちは、国際共同研究により低温電子顕微鏡を用いてタンパク質複合体の立体構造を解析。その結果、CRL4ADDB2ユビキチンリガーゼと結合することでCSNの構造変化が引き起こされ、これによりNEDD8をCUL4Aから取り除くことができるようになることが初めて明らかになりました。CRL4Aが不必要に活性化されるとDDB2自身がユビキチン化を受けて分解されてしまうため、このような制御は細胞のDNA修復活性の維持に重要と言えます。さらに、損傷したDNAとDDB2が結合するとCSNがCRL4ADDB2ユビキチンリガーゼと結合できなくなることが明らかになり、DNA損傷によってユビキチン化が活性化される仕組みが分子レベルで解明されました。
CRL4Aは、DDB2以外にもさまざまな因子と結合することで、多様な機能を担っています。また、CRL4Aを構成する「CUL4A」をはじめとするCullinファミリータンパク質を含むユビキチンリガーゼは200種類以上存在し、いずれもCSNによる制御を受けることが知られています。今回明らかになった制御メカニズムは、普遍的なものと考えられ、DNA修復や皮膚がんの抑制にとどまらず、広範な生物機能の制御においても重要な意義を持つものと考えられます。
CRL4DDB2ユビキチンリガーゼの活性調節機構のモデル